Antibiotika-resistente Bakterien bei Rindern gefunden
Schädliche Bakterien verstecken sich im Viehbestand; traditionelle Methoden finden sie nicht
Die zunehmende Resistenz gegen unsere gängigen Antibiotika ist eine der größten Bedrohungen für die Welt. Da weit verbreitete Bakterien wie Streptokokken und Salmonellen gegen Medikamente resistent werden, können Infektionen, die früher leicht behandelbar waren, jetzt eine schwierige medizinische Herausforderung darstellen.

Neue Forschungen der University of Georgia haben antimikrobiell resistente Salmonellen bei Kühen aufgedeckt.
ndrew Davis Tucker/UGA
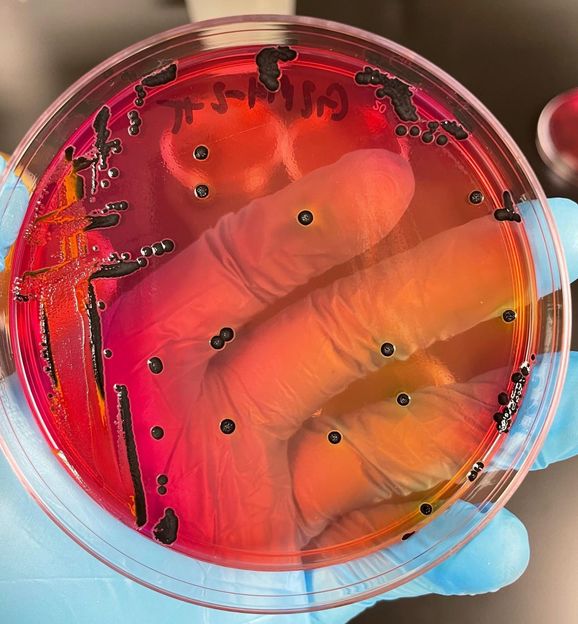
Salmonellenkolonien, die auf Rotindikatorplatten wachsen.
UGA

Neue Forschungsergebnisse der University of Georgia zeigen, dass es möglicherweise mehr antimikrobiell resistente Salmonellen in unseren Lebensmitteln gibt, als Wissenschaftler bisher dachten.
Mit Hilfe der von ihr entwickelten Technologie fanden die UGA-Forscherin Nikki Shariat und Amy Siceloff, eine Doktorandin im ersten Jahr am Fachbereich Mikrobiologie der UGA, heraus, dass herkömmliche Kultivierungsmethoden, die zur Untersuchung von Nutztieren auf problematische Bakterien verwendet werden, häufig arzneimittelresistente Salmonellenstämme übersehen. Diese Erkenntnis hat Auswirkungen auf die Behandlung kranker Nutztiere und der Menschen, die sich durch den Verzehr von kontaminiertem Fleisch infizieren.
Die Studie, die in Antimicrobial Agents and Chemotherapy veröffentlicht wurde, zeigte, dass 60 % der Rinderkotproben mehrere Salmonellenstämme enthielten, die von den traditionellen Testmethoden übersehen wurden. Noch alarmierender ist, dass Shariat herausfand, dass etwa eine von 10 Proben positiv auf einen medikamentenresistenten Salmonellenstamm namens Salmonella Reading getestet wurde. Salmonella Reading ist nicht nur resistent gegen Antibiotika, sondern kann bei Menschen auch schwere Erkrankungen hervorrufen.
Eine neue Technologie taucht auf
Das von Shariat 2015 entwickelte CRISPR-SeroSeq ermöglicht es Forschern, alle in einer Probe vorhandenen Salmonellenarten zu analysieren. Herkömmliche Methoden untersuchen nur eine oder zwei Kolonien von Bakterien und übersehen möglicherweise einige Salmonellenstämme ganz. Shariats Technologie identifiziert molekulare Signaturen in den CRISPR-Regionen der Salmonellen, einem speziellen Teil der Bakterien-DNA. Sie hilft den Forschern auch zu erkennen, welche Bakterienstämme am häufigsten vorkommen.
In der aktuellen Studie fanden Shariat und Kollegen mehrere Salmonellenstämme im Rinderkot, bevor die Tiere mit dem Antibiotikum Tetracyclin behandelt wurden. Nach der Behandlung wurden mehrere der dominanten Salmonellenstämme in der Probe ausgelöscht, wodurch Salmonella Reading gedeihen konnte.
Traditionelle Kultivierungsmethoden übersahen den antibiotikaresistenten Stamm in den ursprünglichen Proben. Erst als das Antibiotikum die häufiger vorkommenden Stämme eliminierte, konnten herkömmliche Methoden Salmonella Reading in den Proben nachweisen.
"Dies deutet darauf hin, dass herkömmliche Tests die Menge an antibiotikaresistenten Bakterien in der Vergangenheit unterschätzt haben", sagte Shariat, ein Assistenzprofessor für Bevölkerungsgesundheit am College of Veterinary Medicine.
Aber CRISPR-SeroSeq ist ein viel empfindlicheres Werkzeug. Es markierte die Salmonella Reading vor der Antibiotika-Behandlung.
"Wir müssen die antimikrobiellen Resistenzprofile der Bakterien kennen, die in den Tieren vorhanden sind", sagte Shariat. "Dieses Wissen könnte dazu führen, dass wir die Art des Antibiotikums, das wir zur Behandlung kranker Tiere verwenden, anders wählen. Es kann uns auch helfen, das beste Antibiotikum für Menschen auszuwählen, die durch den Verzehr von kontaminiertem Fleisch krank werden."
Das Ziel verfehlt
Shariat's Forschung zeigt, dass die derzeitigen Überwachungsmaßnahmen das Ausmaß der antimikrobiellen Resistenz wahrscheinlich unterschätzen.
Agenturen, die antimikrobielle Resistenzen verfolgen, wie die FDA, USDA und CDC, verlassen sich immer noch auf traditionelle Probenahmemethoden, was bedeutet, dass sie möglicherweise Reservoirs von arzneimittelresistenten Bakterien übersehen.
"Das Problem ist, dass man Hunderte von Salmonellenkolonien in einer Probe hat, aber nur ein oder zwei davon zum Testen auswählt", so Shariat. "Es wird zu einem Zahlenspiel, bei dem die Forscher nur die am häufigsten vorkommenden auswählen, und das bedeutet, dass sie die verschiedenen Arten von Salmonellen, die vorhanden sind, unterschätzen."
Die Verwendung von CRISPR-SeroSeq kann dabei helfen, diese Wissenslücke zu schließen und den Forschern eine bessere Vorstellung davon zu geben, wie viele antibiotikaresistente Bakterien vorhanden sind. Diese Informationen können Viehzüchtern helfen, Ausbrüche zu reduzieren und zu kontrollieren, und der Politik Hinweise geben, wie sie gegen eine wachsende Bedrohung der öffentlichen Gesundheit vorgehen kann.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.



































































