Bacterias resistentes a los antibióticos en el ganado
Las bacterias nocivas se esconden en el ganado; los métodos tradicionales no las encuentran
La creciente resistencia a nuestros antibióticos es una de las mayores amenazas a las que se enfrenta el mundo. A medida que bacterias comunes como los estreptococos y la salmonela se hacen resistentes a los medicamentos, lo que antes eran infecciones fácilmente tratables pueden ahora plantear difíciles retos médicos.

Una nueva investigación de la Universidad de Georgia ha descubierto la existencia de salmonela resistente a los antimicrobianos en las vacas.
ndrew Davis Tucker/UGA
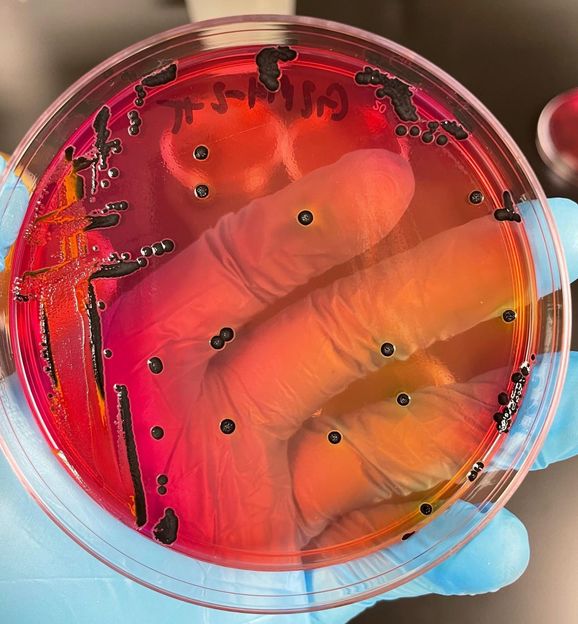
Colonias de Salmonella creciendo en placas indicadoras rojas.
UGA

Una nueva investigación de la Universidad de Georgia muestra que puede haber más salmonela resistente a los antimicrobianos en nuestros animales de abasto de lo que los científicos pensaban.
Gracias a la tecnología que ha desarrollado, la investigadora de la UGA Nikki Shariat y Amy Siceloff, estudiante de primer año de doctorado en el Departamento de Microbiología de la UGA, descubrieron que los métodos de cultivo tradicionales utilizados para analizar el ganado en busca de bacterias problemáticas a menudo pasan por alto las cepas de salmonela resistentes a los medicamentos. Este hallazgo tiene implicaciones para el tratamiento de los animales enfermos y de las personas que se infectan al comer carne contaminada.
El estudio, publicado en Antimicrobial Agents and Chemotherapy, demostró que el 60% de las muestras fecales de ganado contenían múltiples cepas de salmonela que los métodos tradicionales de análisis pasaban por alto. Y lo que es más alarmante, Shariat descubrió que aproximadamente una de cada 10 muestras daba positivo en una cepa de salmonela resistente a los medicamentos, llamada Salmonella Reading. Además de ser resistente a los antibióticos, la Salmonella Reading puede causar enfermedades graves en las personas.
Surge una nueva tecnología
Desarrollado por Shariat en 2015, CRISPR-SeroSeq permite a los investigadores analizar todos los tipos de salmonela presentes en una muestra determinada. Los métodos tradicionales solo examinan una o dos colonias de bacterias, pudiendo pasar por alto algunas cepas de salmonela por completo. La tecnología de Shariat identifica firmas moleculares en las regiones CRISPR de la salmonela, una parte especializada del ADN de la bacteria. También ayuda a los investigadores a identificar qué cepas de la bacteria son más abundantes.
En el estudio actual, Shariat y sus colegas encontraron múltiples cepas de salmonela en las heces del ganado antes de que los animales fueran tratados con el antibiótico tetraciclina. Después del tratamiento, varias de las cepas de salmonela dominantes en la muestra fueron eliminadas, permitiendo que la Salmonella Reading floreciera.
Los métodos de cultivo tradicionales no detectaron la cepa resistente al antibiótico en las muestras originales. Sólo cuando el antibiótico eliminó las cepas más abundantes, los métodos convencionales pudieron detectar la Salmonella Reading en las muestras.
"Esto sugiere que las pruebas tradicionales han subestimado la cantidad de bacterias resistentes a los antibióticos en el pasado", dijo Shariat, profesor asistente de salud de la población en la Facultad de Medicina Veterinaria.
Pero CRISPR-SeroSeq es una herramienta mucho más sensible. Detectó la Salmonella Reading antes del tratamiento con antibióticos.
"Necesitamos conocer los perfiles de resistencia a los antimicrobianos de las bacterias que están presentes en los animales", dijo Shariat. "Ese conocimiento podría hacernos cambiar la elección del tipo de antibiótico que utilizamos para tratar a los animales enfermos. También puede ayudarnos a seleccionar el mejor antibiótico para las personas que enferman por comer carne contaminada."
No dar en el clavo
La investigación de Shariat muestra que los esfuerzos actuales de vigilancia probablemente subestiman la cantidad de resistencia antimicrobiana que existe.
Los organismos que hacen un seguimiento de la resistencia a los antimicrobianos, como la FDA, el USDA y los CDC, entre otros, siguen basándose en métodos de muestreo tradicionales, lo que significa que pueden estar pasando por alto los reservorios de bacterias resistentes a los medicamentos.
"El problema es que hay cientos de colonias de salmonela en una muestra determinada, pero sólo se eligen una o dos para analizarlas", explica Shariat. "Se convierte en un juego de números en el que los investigadores sólo eligen las más abundantes, y esto significa que subestiman los diferentes tipos de salmonela que están presentes".
El uso de CRISPR-SeroSeq puede ayudar a llenar ese vacío de conocimiento, dando a los investigadores una mejor idea de la cantidad de bacterias resistentes a los antibióticos que existen. Esta información puede ayudar a los ganaderos a reducir y controlar los brotes y a orientar la política de lucha contra una amenaza creciente para la salud pública.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.






















































